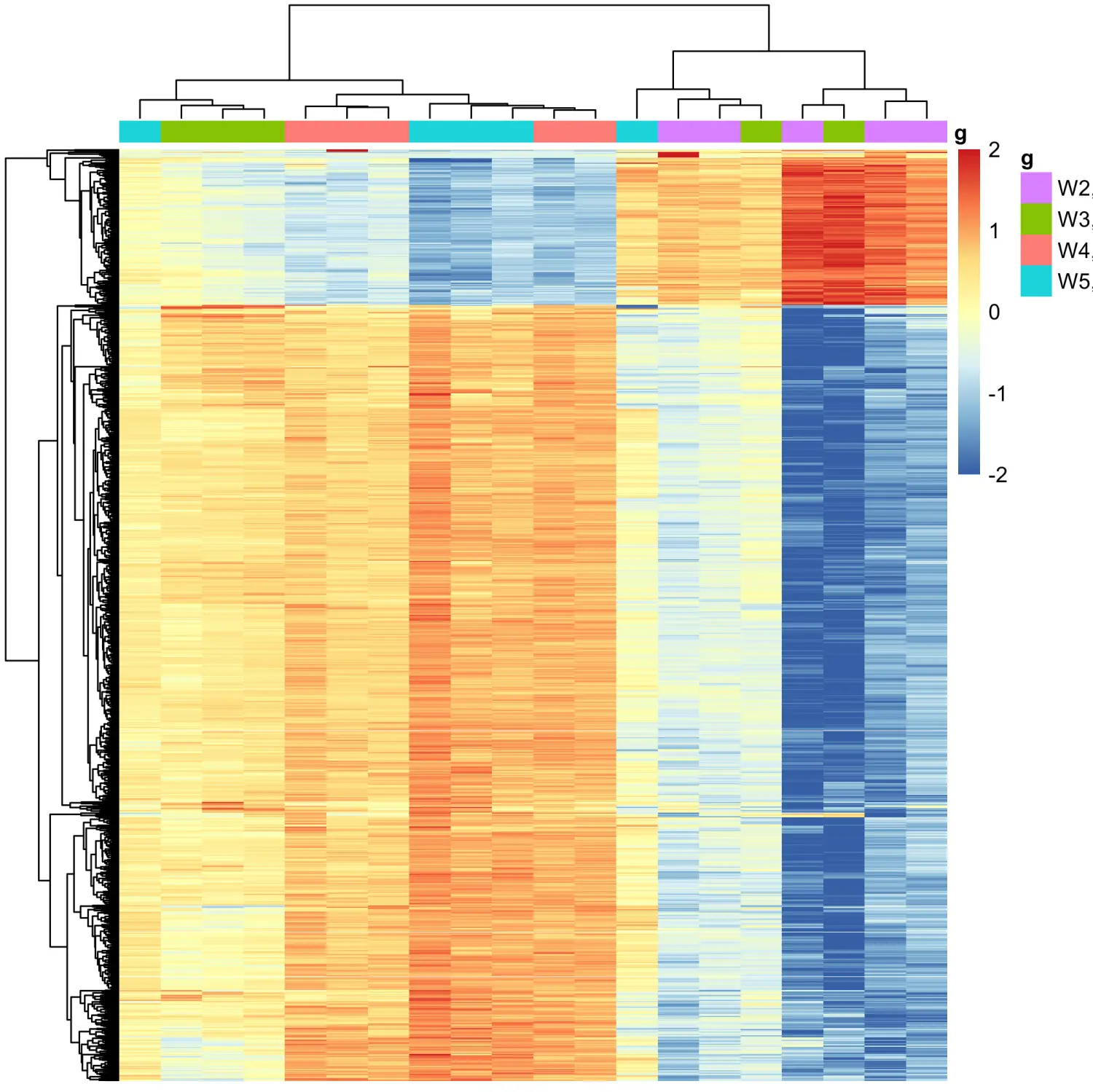
本次配置的环境是Linux版本的R studio server。直接install_github("jmzeng1314/AnnoProbe")安装出现installatio...
-
解决Linux Rstudio server 安装AnnoProbe报错
-
-
独立开发者,终身学习者,喜欢交朋友,欢迎讨论,共同成长,一起进步。
-
@ACh_20d2 根据提供的教程,应该是cor_dist
#' Generates a distance matrix using the square root of (1 - spearman).
#'
#' @param dat.mat Matrix of data (features X samples).
#' @return Distance structure.
#' @export
cor_dist <- function(dat.mat) {
dist.mat <- as.dist(sqrt(1 - cor(dat.mat, method = 'spearman')))
return(dist.mat)
}基于单细胞测序的蛋白活性推断(PISCES)分析流程笔记,凎!
写在前面 感谢califano-lab[https://github.com/califano-lab]团队将代码无私的分享出来=。=! 本流程需要一点点R语言与linux基...
-
请问各位大佬,“RegProcess('pisces-1-net-final.tsv', mat1, out.dir = 'tutorial/', out.name = 'pisces-1-net-')” 这里的mat1是不是 pisces1 <- readRDS('pisces-clust_aracne-mats/pisces-1-arac.rds')?。如果是,在运行过程中连续出现WARNING! NOT CONVERGENT! number of iterations= 1000 是哪里出了问题?
基于单细胞测序的蛋白活性推断(PISCES)分析流程笔记,凎!
写在前面 感谢califano-lab[https://github.com/califano-lab]团队将代码无私的分享出来=。=! 本流程需要一点点R语言与linux基...
-
不看私信<br>看不到的文章是被简书锁了
-
周运来,男,长大了才会遇到的帅哥,稳健,潇洒,大方,靠谱。一段生信缘,一棵技能树。生信技能树核心成员,单细胞天地供稿人,简书创作者,单细胞数据科学家。
-
公众号:TOP生物信息<br>B站:TOP菌<br>WeChat:TOPJUN996<br><br>top2读研,学习方向:癌症基因组、肿瘤免疫、单细胞组学
-
生信阿拉丁,为实用而生
-
-
-
-
-
收集一些生物信息学的分析流程的文章。感谢这些简友整理的生信分析流程! 如果简友投稿没有被我收入专题,可能是我没看到或者收入到其他针对性更强的专题中了。望见谅! 🕛🕐🕑🕒🕓🕔🕕🕖🕗🕘🕙🕚 文章的收录是有顺序的,查看的时候请点击“最新收录”然后进行查看。 👣 全基因组测序(WGS)分析 👣 转录组分析RNA-seq的分析流程 👣 染色质免疫共沉淀技术ATAC-Seq、ChIP-Seq分析 👣 单细胞scRNA-seq分析 👣 癌症的预后标志物查找 👣 系统发育演化进化树构建与分析 👣 群体遗传学分析 👣 全基因组关联分析(GWAS) 👣 基因家族的分析 👣 甲基化测序与分析 👣 KEGG/GO功能富集分析 👣 图形绘制具体细节的比如热图、曼哈顿图的绘制等 👣 python、perl、R的包的使用过程 👣 具体工具的使用,比如GATK的使用过程 👣 数据库的挖掘方法,比如TCGA数据库的使用 👣 另外就是整个文章走下来的流程 👣 👣 👣
-
从0开始,包括软件安装,环境设置,原始数据下载,转换,差异基因分析等整个流程
-
中科院上海生科院硕博连读生。<br>
-
生信技能树,生信菜鸟团,jimmy